AutoDock Vina: Różnice pomiędzy wersjami
| Linia 1: | Linia 1: | ||
| − | + | <small>< [[Podręcznik użytkownika KDM]] < [[Oprogramowanie KDM]] < [[Oprogramowanie naukowe]]</small> | |
| − | + | {{aplikacja|nazwa=AutoDock Vina|logo=|serwer=[[Nova]]|wersja=1}} | |
| − | + | '''AutoDock Vina''' - program wykorzystywany do dokowania molekularnego oraz screeningu układów białko-ligand. Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (''scoring function'') oraz wprowadzenie możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x). | |
| − | Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (''scoring function'') oraz wprowadzenie | ||
| − | możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości | ||
| − | uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x). | ||
{| | {| | ||
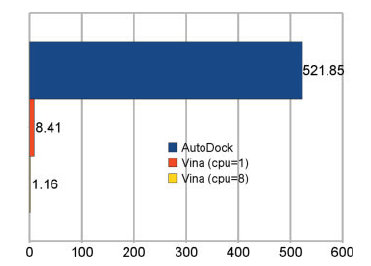
| − | | [[File:Speed_autodock_vs_vina.png|thumb| upright=2|Średni czas w minutach na statystyczny kompleks. 2 x Intel Xeon @ 2.66 (2 x 4 rdzenie).]] | + | | [[File:Speed_autodock_vs_vina.png|thumb| upright=2|Średni czas obliczeń w minutach na statystyczny kompleks. 2 x Intel Xeon @ 2.66 (2 x 4 rdzenie).]] |
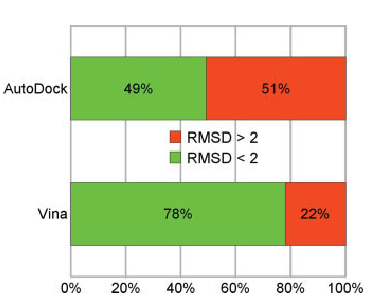
| [[File:Rmsd_autodock_vina.png|thumb|upright=1.81|Porównanie jakości wyników AutoDock 4.0.x względem AutoDock Vina.]] | | [[File:Rmsd_autodock_vina.png|thumb|upright=1.81|Porównanie jakości wyników AutoDock 4.0.x względem AutoDock Vina.]] | ||
|} | |} | ||
| − | + | === Przepływ danych === | |
| + | Na każdym etapie można zastosować alternatywne oprogramowanie. | ||
| − | Przygotowanie pliku wsadowego w '''MGLTools''' (*.PDBQT). | + | *Przygotowanie pliku wsadowego w '''MGLTools''' (*.PDBQT). |
| − | *przygotowanie struktury białka (dodanie wodorów, określenie rozmiaru przestrzeni dopasowania) | + | **przygotowanie struktury białka (dodanie wodorów, określenie rozmiaru przestrzeni dopasowania) |
| − | *przygotowanie ligandu (określenie wiązań | + | **przygotowanie ligandu (określenie wiązań rotacyjnych) |
| − | Przeprowadzenie obliczeń w '''AutoDock Vina'''. | + | *Przeprowadzenie obliczeń w '''AutoDock Vina''' w WCSS. |
| + | **AutoDock Vina uruchamiany jest skryptem <code>sub-autodock-vina</code> | ||
| − | * | + | *Interpretacja wyników w '''PyMOL'''. |
| − | + | === Licencja === | |
| + | Program udostępniony jest na licencji Apache, z kilkoma zastrzeżeniami co do użytku komercyjnego, niekomercyjnego i prac wywiedzionych ([http://vina.scripps.edu/LICENSE tekst licencji]) | ||
| − | + | W publikacjach, w których do liczenia korzystano z AutoDock Vina, należy cytować artykuł: "O. Trott, A. J. Olson, AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading, Journal of Computational Chemistry 31 (2010) 455-461" | |
| − | + | === Linki zewnętrzne === | |
| − | + | * [http://vina.scripps.edu Strona domowa programu Autodock Vina] | |
| − | |||
| − | + | [[Kategoria:Oprogramowanie]] | |
| + | [[Kategoria:Podręcznik użytkownika]] | ||
Wersja z 10:19, 16 sie 2011
< Podręcznik użytkownika KDM < Oprogramowanie KDM < Oprogramowanie naukowe
| AutoDock Vina | |
|---|---|
| Serwer | Wersja |
| Nova | 1 |
| Kontakt | |
| kdm@wcss.pl | |
AutoDock Vina - program wykorzystywany do dokowania molekularnego oraz screeningu układów białko-ligand. Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (scoring function) oraz wprowadzenie możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x).
Przepływ danych
Na każdym etapie można zastosować alternatywne oprogramowanie.
- Przygotowanie pliku wsadowego w MGLTools (*.PDBQT).
- przygotowanie struktury białka (dodanie wodorów, określenie rozmiaru przestrzeni dopasowania)
- przygotowanie ligandu (określenie wiązań rotacyjnych)
- Przeprowadzenie obliczeń w AutoDock Vina w WCSS.
- AutoDock Vina uruchamiany jest skryptem
sub-autodock-vina
- AutoDock Vina uruchamiany jest skryptem
- Interpretacja wyników w PyMOL.
Licencja
Program udostępniony jest na licencji Apache, z kilkoma zastrzeżeniami co do użytku komercyjnego, niekomercyjnego i prac wywiedzionych (tekst licencji)
W publikacjach, w których do liczenia korzystano z AutoDock Vina, należy cytować artykuł: "O. Trott, A. J. Olson, AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading, Journal of Computational Chemistry 31 (2010) 455-461"