AutoDock Vina: Różnice pomiędzy wersjami
| Linia 1: | Linia 1: | ||
<small>< [[Podręcznik użytkownika KDM]] < [[Oprogramowanie KDM]] < [[Oprogramowanie naukowe]]</small> | <small>< [[Podręcznik użytkownika KDM]] < [[Oprogramowanie KDM]] < [[Oprogramowanie naukowe]]</small> | ||
| − | {{aplikacja|nazwa=AutoDock Vina|logo=|serwer=[[Nova]]|wersja=1}} | + | {{aplikacja|nazwa=AutoDock Vina|logo=|serwer=[[Nova]]|wersja=1.1.2}} |
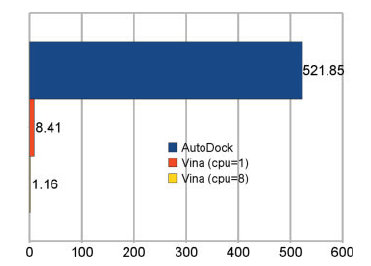
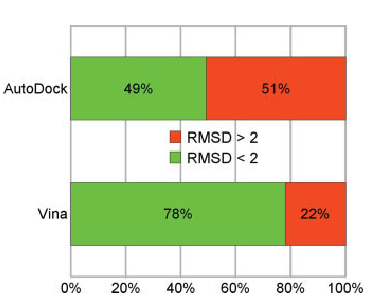
'''AutoDock Vina''' - program wykorzystywany do dokowania molekularnego oraz screeningu układów białko-ligand. Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (''scoring function'') oraz wprowadzenie możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x). | '''AutoDock Vina''' - program wykorzystywany do dokowania molekularnego oraz screeningu układów białko-ligand. Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (''scoring function'') oraz wprowadzenie możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x). | ||
| Linia 21: | Linia 21: | ||
*Interpretacja wyników w '''PyMOL'''. | *Interpretacja wyników w '''PyMOL'''. | ||
| + | |||
| + | |||
| + | ===Korzystanie w WCSS=== | ||
| + | Uruchamiamy skryptem "sub-autodock_vina", ograniczeni na jeden węzeł (12 CPU), skrypt domyślnie rezerwuje 1800mb per jeden rdzeń. | ||
| + | sub-autodock_vina | ||
| + | Sposob uzycia: [plik_konfiguracyjny] [liczba_rdzeni] [kolejka] | ||
=== Licencja === | === Licencja === | ||
Wersja z 15:44, 16 sie 2011
< Podręcznik użytkownika KDM < Oprogramowanie KDM < Oprogramowanie naukowe
| AutoDock Vina | |
|---|---|
| Serwer | Wersja |
| Nova | 1.1.2 |
| Kontakt | |
| kdm@wcss.pl | |
AutoDock Vina - program wykorzystywany do dokowania molekularnego oraz screeningu układów białko-ligand. Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (scoring function) oraz wprowadzenie możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x).
Przepływ danych
Na każdym etapie można zastosować alternatywne oprogramowanie.
- Przygotowanie pliku wsadowego w MGLTools (*.PDBQT).
- przygotowanie struktury białka (dodanie wodorów, określenie rozmiaru przestrzeni dopasowania)
- przygotowanie ligandu (określenie wiązań rotacyjnych)
- Przeprowadzenie obliczeń w AutoDock Vina w WCSS.
- AutoDock Vina uruchamiany jest skryptem
sub-autodock-vina
- AutoDock Vina uruchamiany jest skryptem
- Interpretacja wyników w PyMOL.
Korzystanie w WCSS
Uruchamiamy skryptem "sub-autodock_vina", ograniczeni na jeden węzeł (12 CPU), skrypt domyślnie rezerwuje 1800mb per jeden rdzeń.
sub-autodock_vina Sposob uzycia: [plik_konfiguracyjny] [liczba_rdzeni] [kolejka]
Licencja
Program udostępniony jest na licencji Apache, z kilkoma zastrzeżeniami co do użytku komercyjnego, niekomercyjnego i prac wywiedzionych (tekst licencji)
W publikacjach, w których do liczenia korzystano z AutoDock Vina, należy cytować artykuł: "O. Trott, A. J. Olson, AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading, Journal of Computational Chemistry 31 (2010) 455-461"
Linki zewnętrzne
| Oprogramowanie naukowe |
Abaqus ⋅ ABINIT ⋅ ADF ⋅ Amber ⋅ ANSYS [ ANSYS CFD: Fluent, CFX, ICEM; Mechanical ] ⋅ AutoDock ⋅ BAGEL ⋅ Beast ⋅ Biovia [ Materials Studio, Discovery Studio ] ⋅ Cfour ⋅ Comsol ⋅ CP2K ⋅ CPMD ⋅ CRYSTAL ⋅ Dalton ⋅ Dask ⋅ DIRAC ⋅ FDS-SMV ⋅ GAMESS ⋅ Gaussian ⋅ Gromacs ⋅ IDL ⋅ Lumerical [ FDTD, MODE ] ⋅ Mathcad ⋅ Mathematica⋅ Matlab ⋅ Molcas ⋅ Molden ⋅ Molpro ⋅ MOPAC ⋅ NAMD ⋅ NBO ⋅ NWChem ⋅ OpenFOAM ⋅ OpenMolcas ⋅ Orca ⋅ Quantum ESPRESSO ⋅ R ⋅ Rosetta ⋅ SIESTA ⋅ Tinker ⋅ TURBOMOLE ⋅ VASP ⋅ VMD ⋅ WIEN2k |
|---|