AutoDock Vina: Różnice pomiędzy wersjami
| (Nie pokazano 28 wersji utworzonych przez 4 użytkowników) | |||
| Linia 1: | Linia 1: | ||
| − | + | <small>< [[Podręcznik użytkownika KDM]] < [[Oprogramowanie KDM]] < [[Oprogramowanie naukowe]]</small> | |
| − | + | '''AutoDock Vina''' - program wykorzystywany do dokowania molekularnego oraz screeningu układów białko-ligand. Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (''scoring function'') oraz wprowadzenie możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x). | |
| − | Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (''scoring function'')oraz wprowadzenie | ||
| − | możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości | ||
| − | uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x). | ||
| − | |||
{| | {| | ||
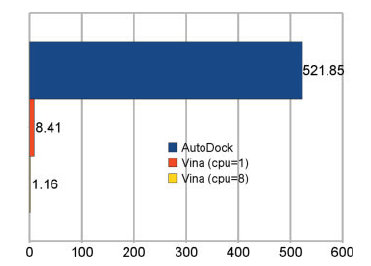
| − | | [[File:Speed_autodock_vs_vina.png|thumb| upright=2|Średni czas w minutach na statystyczny kompleks. 2 x Intel Xeon @ 2.66 (2 x 4 rdzenie). | + | | [[File:Speed_autodock_vs_vina.png|thumb| upright=2|Średni czas obliczeń w minutach na statystyczny kompleks. 2 x Intel Xeon @ 2.66 (2 x 4 rdzenie).]] |
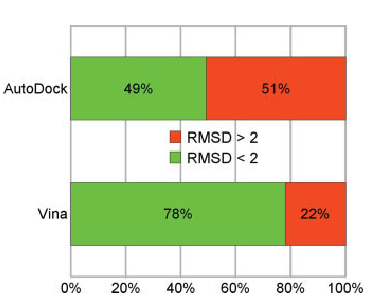
| − | | [[File:Rmsd_autodock_vina.png|thumb|upright=1.81|Porównanie jakości wyników AutoDock 4.0.x względem AutoDock Vina | + | | [[File:Rmsd_autodock_vina.png|thumb|upright=1.81|Porównanie jakości wyników AutoDock 4.0.x względem AutoDock Vina.]] |
|} | |} | ||
| − | + | === Przepływ danych === | |
| + | Na każdym etapie można zastosować alternatywne oprogramowanie. | ||
| − | Przygotowanie pliku wsadowego w '''MGLTools''' (*.PDBQT). | + | *Przygotowanie pliku wsadowego w '''MGLTools''' (*.PDBQT). |
| − | *przygotowanie struktury białka (dodanie wodorów, określenie rozmiaru przestrzeni dopasowania) | + | **przygotowanie struktury białka (dodanie wodorów, określenie rozmiaru przestrzeni dopasowania) |
| − | *przygotowanie ligandu (określenie wiązań | + | **przygotowanie ligandu (określenie wiązań rotacyjnych) |
| − | Przeprowadzenie obliczeń w '''AutoDock Vina'''. | + | *Przeprowadzenie obliczeń w '''AutoDock Vina''' w WCSS. |
| + | **AutoDock Vina uruchamiany jest skryptem <code>sub-autodock_vina</code> | ||
| − | * | + | *Interpretacja wyników w '''PyMOL''', '''ADT''' lub innej aplikacji do tego przeznaczonej. |
| − | + | ===Korzystanie w WCSS=== | |
| + | Uruchamiamy skryptem "sub-autodock_vina" - ograniczenie na jeden węzeł (12 CPU). Skrypt domyślnie rezerwuje 1800mb per jeden rdzeń. | ||
| + | sub-autodock_vina | ||
| + | Sposob uzycia: [plik_konfiguracyjny] [liczba_rdzeni] [kolejka] | ||
| − | + | ===Przyklad pliku konfiguracyjnego=== | |
| − | + | receptor = bialko.pdbqt # nazwa pliku z przygotowaną strukturą receptora (może byc ścieżka) | |
| − | + | ligand = ligand.pdbqt # nazwa pliku z przygotowaną strukturą liganda (może byc ścieżka) | |
| − | + | center_x = 11 # współrzędna x-owa środka przestrzeni dopasowania | |
| + | center_y = 90.5 # współrzędna y-owa środka przestrzeni dopasowania | ||
| + | center_z = 57.5 # współrzędna z-owa środka przestrzeni dopasowania | ||
| + | size_x = 22 # składowa x-owa długości przestrzeni dopasowania w Å | ||
| + | size_y = 24 # składowa y-owa długości przestrzeni dopasowania w Å | ||
| + | size_z = 28 # składowa z-owa długości przestrzeni dopasowania w Å | ||
| + | exhaustiveness = 16 # ilość iteracji | ||
| − | W publikacjach, w których do liczenia korzystano z AutoDock Vina, | + | === Licencja === |
| − | + | Program udostępniony jest na licencji Apache, z kilkoma zastrzeżeniami co do użytku komercyjnego, niekomercyjnego i prac wywiedzionych ([http://vina.scripps.edu/LICENSE tekst licencji]) | |
| − | efficient optimization and multithreading, Journal of Computational Chemistry 31 (2010) 455-461" | + | |
| + | W publikacjach, w których do liczenia korzystano z AutoDock Vina, należy cytować artykuł: "O. Trott, A. J. Olson, AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading, Journal of Computational Chemistry 31 (2010) 455-461" | ||
| + | === Informacje o wykorzystaniu === | ||
| + | {{Podziękowanie_WCSS}} | ||
| + | === Linki zewnętrzne === | ||
| + | * [http://vina.scripps.edu Strona domowa programu Autodock Vina] | ||
| + | |||
| + | {{oprogramowanie}} | ||
| + | |||
| + | [[Kategoria:Oprogramowanie obecnie niewspierane]] | ||
| + | [[Kategoria:Podręcznik użytkownika]] | ||
Aktualna wersja na dzień 13:15, 1 mar 2016
< Podręcznik użytkownika KDM < Oprogramowanie KDM < Oprogramowanie naukowe
AutoDock Vina - program wykorzystywany do dokowania molekularnego oraz screeningu układów białko-ligand. Implementacja nowych algorytmów optymalizacji, funkcji dopasowania (scoring function) oraz wprowadzenie możliwości zrównoleglenia obliczeń zaowocowało wynikami lepszej jakości uzyskanymi w do 2 rzędów krótszym czasie względem poprzedniej generacji oprogramowania (AutoDock4.x).
Przepływ danych
Na każdym etapie można zastosować alternatywne oprogramowanie.
- Przygotowanie pliku wsadowego w MGLTools (*.PDBQT).
- przygotowanie struktury białka (dodanie wodorów, określenie rozmiaru przestrzeni dopasowania)
- przygotowanie ligandu (określenie wiązań rotacyjnych)
- Przeprowadzenie obliczeń w AutoDock Vina w WCSS.
- AutoDock Vina uruchamiany jest skryptem
sub-autodock_vina
- AutoDock Vina uruchamiany jest skryptem
- Interpretacja wyników w PyMOL, ADT lub innej aplikacji do tego przeznaczonej.
Korzystanie w WCSS
Uruchamiamy skryptem "sub-autodock_vina" - ograniczenie na jeden węzeł (12 CPU). Skrypt domyślnie rezerwuje 1800mb per jeden rdzeń.
sub-autodock_vina Sposob uzycia: [plik_konfiguracyjny] [liczba_rdzeni] [kolejka]
Przyklad pliku konfiguracyjnego
receptor = bialko.pdbqt # nazwa pliku z przygotowaną strukturą receptora (może byc ścieżka) ligand = ligand.pdbqt # nazwa pliku z przygotowaną strukturą liganda (może byc ścieżka) center_x = 11 # współrzędna x-owa środka przestrzeni dopasowania center_y = 90.5 # współrzędna y-owa środka przestrzeni dopasowania center_z = 57.5 # współrzędna z-owa środka przestrzeni dopasowania size_x = 22 # składowa x-owa długości przestrzeni dopasowania w Å size_y = 24 # składowa y-owa długości przestrzeni dopasowania w Å size_z = 28 # składowa z-owa długości przestrzeni dopasowania w Å exhaustiveness = 16 # ilość iteracji
Licencja
Program udostępniony jest na licencji Apache, z kilkoma zastrzeżeniami co do użytku komercyjnego, niekomercyjnego i prac wywiedzionych (tekst licencji)
W publikacjach, w których do liczenia korzystano z AutoDock Vina, należy cytować artykuł: "O. Trott, A. J. Olson, AutoDock Vina: improving the speed and accuracy of docking with a new scoring function, efficient optimization and multithreading, Journal of Computational Chemistry 31 (2010) 455-461"
Informacje o wykorzystaniu
Wszelkie publikacje, (w tym prace doktorskie i dyplomowe) wykorzystujące wyniki obliczeń wykonanych na komputerach WCSS, powinny zawierać podziękowania postaci (odpowiednio do języka publikacji):
"Obliczenia wykonano na komputerach Wrocławskiego Centrum Sieciowo-Superkomputerowego (http://www.wcss.pl), grant obliczeniowy Nr ... "
"Calculations have been carried out in Wroclaw Centre for Networking and Supercomputing (http://www.wcss.pl), grant No. ..."
Linki zewnętrzne
| Oprogramowanie naukowe |
Abaqus ⋅ ABINIT ⋅ ADF ⋅ Amber ⋅ ANSYS [ ANSYS CFD: Fluent, CFX, ICEM; Mechanical ] ⋅ AutoDock ⋅ BAGEL ⋅ Beast ⋅ Biovia [ Materials Studio, Discovery Studio ] ⋅ Cfour ⋅ Comsol ⋅ CP2K ⋅ CPMD ⋅ CRYSTAL ⋅ Dalton ⋅ Dask ⋅ DIRAC ⋅ FDS-SMV ⋅ GAMESS ⋅ Gaussian ⋅ Gromacs ⋅ IDL ⋅ Lumerical [ FDTD, MODE ] ⋅ Mathcad ⋅ Mathematica⋅ Matlab ⋅ Molcas ⋅ Molden ⋅ Molpro ⋅ MOPAC ⋅ NAMD ⋅ NBO ⋅ NWChem ⋅ OpenFOAM ⋅ OpenMolcas ⋅ Orca ⋅ Quantum ESPRESSO ⋅ R ⋅ Rosetta ⋅ SIESTA ⋅ Tinker ⋅ TURBOMOLE ⋅ VASP ⋅ VMD ⋅ WIEN2k |
|---|